

このページにはプロモーションが含まれています。
忙しくても利用しやすい理系学生・院生特化型就活サイト
qPCRのインターナルコントロールの設定に便利なNormFinderの使い方マニュアル
リアルタイムqPCRの内部標準としてどの遺伝子を使うべきか?
GAPDHやβ-actin、HPRTなどのハウスキーピング遺伝子を使うのが一般的だと思います。
ですが、サンプルの種類や状態によっては、これらの遺伝子発現も結構ぶれてしまって、判断に迷う場合があります。
最適なインターナルコントロールを選択するのに使われるソフトがNormFinderです。
https://moma.dk/normfinder-software
他にもGeNormとかもありますが、NormFinderはエクセルのアドインとして無料で配布されていますので、使いやすいと思います。
ところが、NormFinderの使い方を日本語で解説したものが見つからなかったので、日本語マニュアルを作ってみました。
エクセルのアドインとRがありますが、Rを使ったことのない人はエクセル版をお勧めします。
まず、NormFinderのサイトから「NormFinder Excel Add-In」をダウンロードし、エクセルのアドインとしてインストールします。
Excel 2003用となっていますが、2010でも一応使えました。
アドインのインストール方法はこちらなどを参考にしてみてください。
画像のようにエクセルで遺伝子発現量の表を作ります。
1行目はサンプル名を(適当でいいと思います)
最終行はグループの数字(整数でなければなりません)
1列目には遺伝子名を入力します。
各セルには、遺伝子発現量(Ct値ではない)を入力します。
プライマーの増幅効率もリファレンス遺伝子の決定に影響を与える因子ですので、DDCt法よりは検量線法で決定した遺伝子発現量のほうが良いでしょう。
また、このソフトは、3つ以上のインターナルコントロール候補からベストなものを選ぶためのアドインなので、遺伝子は3つ以上必要です。
例として、Gene A,B,Cの3つの遺伝子発現の表と作りました。
6-wellディッシュから6つのmRNAサンプルを回収する作業を3回の繰り返しています。
したがって、サンプル数は6*3=18です。
それぞれの繰り返し実験に1,2,3の番号を付け、別々のグループということにしています(最終行)
メニューの「アドイン」から「NormFinder」を起動します。
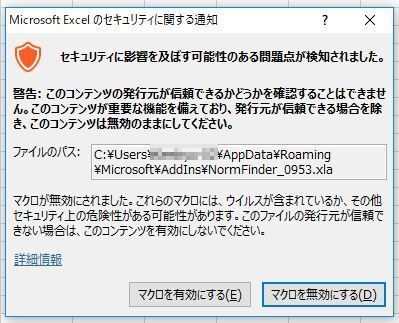
「マクロを有効にする」とクリック。

「Accept」をクリック。
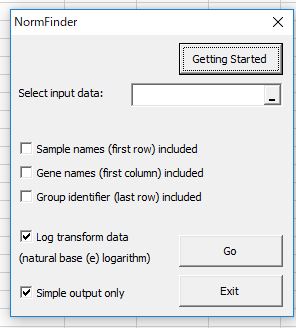
上のようなボックスが出ます。
Select input dataで、表のセル全体(見出しも含む)を選択します。
その下の3つのチェックボックスは、必要に応じてチェックを外します。
グループが1つの場合は、3つ目のチェックボックスは不要ですね。
「Log transform data」はチェックを外します。
「Simple output only」は外しても構いませんが、付けておいて損はしません。
このような感じで結果が出ます。
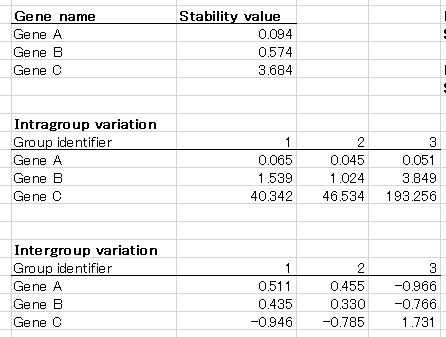
「Stability value」が低いほど発現量が安定しているということになります。
Intragroup variationやIntergroup variationは参考情報で、あまり気にしなくても良いと思います。
さて、どの程度のstability valueであればインターネルコントロールとしてOKなのか、という点ですが、次の論文が参考になると思います。
http://journals.plos.org/plosone/article?id=10.1371/journal.pone.0122515
Fig2Aや2Cを参考にすると、最も安定なもので0.1程度、不安定なもので0.6-0.9程度になっています。
GeNormだと0.15以下というのが一応の目安という話もあります。
上記の例だと、圧倒的にGene A一択ですね。
Gene Bも良くありません。
Gene Cは問題外ですね。
以上です。
リアルタイムqPCRのインターナルコントロールにどの遺伝子を使えばよいか困っている人、NormFinderの使い方を知りたいという方の参考になりましたら幸いです。
